
Projektanslag 2015
Evolution of new genes and proteins
Huvudsökande:
Dan Andersson, professor i medicinsk bakteriologi
Medsökande:
Leif Andersson
Siv Andersson
Johan Åqvist
Per Jemth
Maria Selmer
Lärosäte:
Uppsala universitet
Beviljat anslag:
46,7 miljoner kronor under fem år
– På lång sikt kan vår forskning till exempel ha betydelse för utveckling av nya behandlingar mot sjukdom med genetisk bakgrund eller mot virus och bakterier. Men det egentliga målet med projektet är att på en grundläggande nivå förstå mekanismerna bakom hur nya gener och proteiner uppstår och utvecklas, säger professor Dan I Andersson, Uppsala universitet.
Det är framför allt två olika typer av mekanismer som står i fokus för forskarnas intresse: när nya gener med nya funktioner uppstår från existerande gener, samt evolution av nya gener och proteiner från slumpmässiga DNA-sekvenser som inte har någon likhet med existerande gener och proteiner.
Den första mekanismen kallas duplikation-divergens-modellen. Den beskriver hur en gen som kodar för ett protein eller en peptid med huvudfunktionen A, men också för en svag sidofunktion B, kan utvecklas till två olika gener. Om sidofunktionen B blir av värde för cellen så kan genen dupliceras och efter att mutationer uppstått i en av kopiorna som gör att B-funktionen förbättrats så har cellen två kopior av genen – en som utför A-funktionen väl och en som utför B-funktionen väl. Det här är en standardmodell för hur en ny gen uppstår som är väl beskriven i naturliga system.
– Duplikation-divergens-modellen har min forskargrupp redan arbetat med i tio år och nu går vi vidare för att bättre förstå detaljerna. I denna del av projektet vet vi ganska säkert att vi kommer att få resultat.
Det som gör Dan I Andersson mest entusiastisk är ändå den andra typen av mekanism, det vill säga evolution av nya gener och proteiner från slumpmässiga DNA-sekvenser. Det är ganska enkelt att förstå att när man redan har någonting, det vill säga en gen som kodar för ett protein med en funktion, så kan det modifieras och få en ny funktion. Men hur går man från ”ingenting”, det vill säga slumpmässiga DNA-sekvenser, till en funktion som innebär en liten fördel och därmed kan gynnas av det naturliga urvalet?
– För mig är det en central fråga i projektet och evolutionsbiologins heliga graal, för idag har ingen en aning om hur det går till. I denna del av projektet är också risken större, för det är inte säkert att vi kommer att hitta några slumpmässiga peptider som kan utvecklas till riktiga, funktionella enzym.
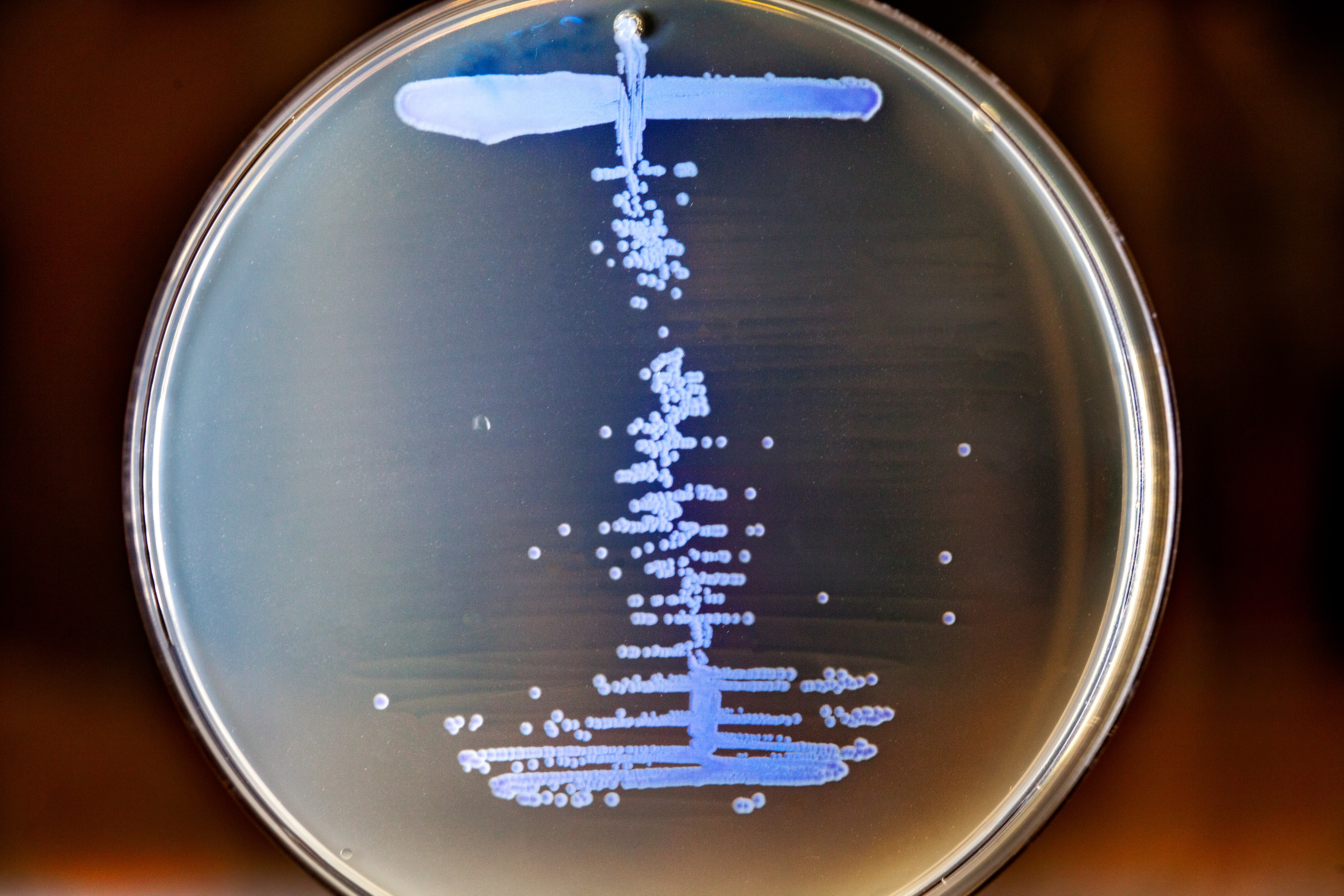
Jättebibliotek
Med hjälp av evolutionsexperiment i laboratoriemiljö, kombinerat med struktur- och funktionsstudier och bioinformatiska metoder, kommer forskarna att analysera hur nya gener uppstår i realtid i levande organismer som till exempel bakterier. Deras råmaterial är bland annat ett jättebibliotek med cirka 100 miljarder slumpmässiga gensekvenser. I detta material försöker de först identifiera sekvenser som har en biologisk funktion. De som hittas undersöks sedan vidare med avseende på struktur och funktion.
– Vi tar slumpmässiga gensekvenser, sätter dem på en plasmid, överuttrycker dem, och studerar sedan om de ger bakterien en speciell fenotyp, det vill säga en viss specifik egenskap. Kan de till exempel rädda mutanter som inte kan göra vissa aminosyror eller andra saker som de behöver?
Ett antagande som forskarna utgår ifrån är att sådana här små skillnader i aktiviteter inte är så sällsynta som man hittills har trott, utan att det faktiskt är en väsentlig del av alla varianter som kan ge en liten biologisk funktion som sedan kan utvecklas till något som blir bättre och bättre. Utifrån en sådan startpunkt kan forskarna sedan ställa en mängd frågor, till exempel: Finns det många utvecklingsvägar mot ett riktigt, normalt enzym? Eller är möjligheterna begränsade, det vill säga att utvecklingen följer vissa bestämda vägar?
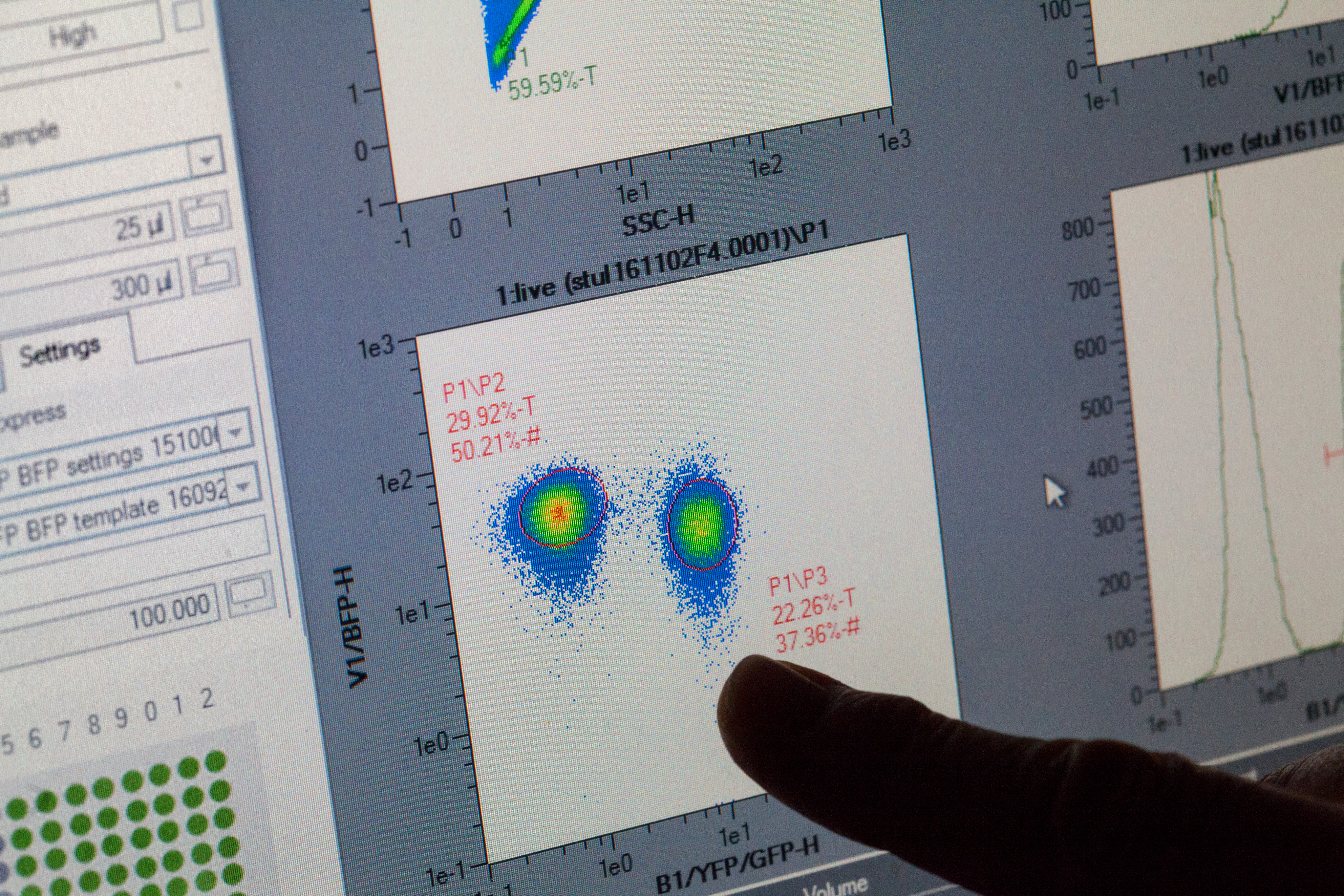
Tvärvetenskaplig forskargrupp
Forskarna som deltar i projektet har väsentligt olika bakgrund: tre av forskningsledarna är biologer, en är strukturbiolog, en är biokemist och en är beräkningskemist. Dan I Andersson menar att det är den sammanlagda mängden kompetenser som är projektets styrka.
– Jag tycker det är en fantastiskt spännande konstellation av människor som ingår i det här projektet. Tillsammans har vi kapaciteten att förstå hur nya gener och nya funktionella proteiner uppstår och utvecklas i detalj. Det här är också en grupp som fungerar väldigt bra tillsammans. Dessutom sitter vi nära varandra på Uppsala biomedicinska centrum, vilket är väldigt värdefullt.
Varför är den här forskningen kul?
– Det är känslan att vara först. Vad jag vet så har ingen tidigare testat det här angreppssättet i en så stor skala som vi gör nu, i alla fall har ingen publicerat resultat från sådana studier. Dessutom handlar det här projektet om en fråga som känns som den mest fundamentala som jag har sysslat med under hela min karriär.
Text Anders Esselin
Bild Magnus Bergström
Ordförklaringar
Peptider är molekyler som består av en kedja av aminosyror. De skiljer sig från proteiner genom att de är mindre. Gränsen mellan en peptid och ett protein brukar dras vid 50 aminosyror.
Enzymer är proteiner som på ett eller annat sätt påverkar hastigheten på kemiska reaktioner.
Plasmider är ringformade DNA-molekyler med en förhållandevis liten mängd gener som förekommer i bakterier.
Mer om Dan I. Anderssons forskning
Nya snabbtester lovande verktyg i kampen mot antibiotikaresistens



